Colectarea probelor de țesut:
Pentru analiza diversităţii genetice la nivel de ADN a reproducătorilor sălbatici de sturioni, a puilor crescuți pentru popularea Dunării şi a puilor si juvenililor sălbatici capturați din mare în cursul pescuitului de evaluare sau accidental în pescuitul costier din țările de la Marea Neagră, au fost colectate probe de țesut.
Probele de ţesut s-au recoltat prin rezecția unui fragment de cca. 1 cm2 din înotătoarea anală, care s-au conservat în tuburi de 5 ml in etanol 99,5%.

Procedeul de colectare pentru analize genetice / ADN a fragmentului din înotătoarea anală la un pui de nisetru(foto original R.Suciu)
Datele de distribuție și probele de țesut au fost obținute prin pescuit experimental în perioada 2014-2015 în zona de vărsare a brațului Sfântu Gheorghe, Sahalin-Zătoane, și prin colaborarea în cadrul proiectului cu experții străini din Ucraina și Georgia. Au fost incluse în studiu un număr de 500 de probe de țesut.
Pentru prima dată diversitatea genetică a unui număr mare de exemplare de sturioni capturate în bazinul Mării Negre a fost investigată prin secvențierea regiunii D-loop, metoda care identifică la nivel de nucleotide variabilitatea genetica la nivel populațional.
Extracția și purificarea ADN-ului din probele de lichid analizate
Pentru extracția ADN-ului, fiecare proba de țesut a fost in prealabil spălata cu apă bidistilată, uscată și triturată mecanic.
Extracţia și purificarea ADN-ului a fost realizata folosind NucleoSpin® Food (MACHEREY-NAGEL) , în conformitate cu recomandările producătorului.

Etapa de pregătire a probelor de țesut de sturioni în vederea extracției ADN-ului(foto original R.Suciu)
Kitul de extracție a ADN-ului din probele de țesut de sturioni utilizat(foto original R.Suciu)

Etapa de lucru în timpul extracției de ADN din probele de țesut de sturioni(foto original R.Suciu)
ADN-ul total a fost extras de la peste 500 de probe. Calitatea ADN-ului depinde foarte mult de condițiile de colectare și conservare în alcool. Se observă o diferență mare de calitate între probele colectate de la pui de sturioni şi sub-adulți. Astfel, probele subțiri (pui) sunt mult mai eficient fixate comparativ cu cele de grosime medie sau mare (sub-adulți).
Pentru remedierea acestui inconvenient, este necesara colectarea țesuturilor cât mai subțiri (vârfuri de înotătoare eliminând zona mortificată), șterse cu alcool pentru eliminarea mucusului ce conține o mare varietate de enzime ce produc degradarea ADN-ului, şi conservarea fragmentului în volum cât mai mare de alcool. Au fost obținute probe de ADN degradate și în cazul celor prelevate în timpul verii, când temperatura ambientala era foarte ridicată.
Degradarea ADN-ului se evidențiază electroforetic, sub forma unor dâre, formate dintr-o multitudine de fragmente de ADN cu dimensiuni variabile. Datorită degradării, analiza acestor probe este dificilă, uneori chiar imposibilă.
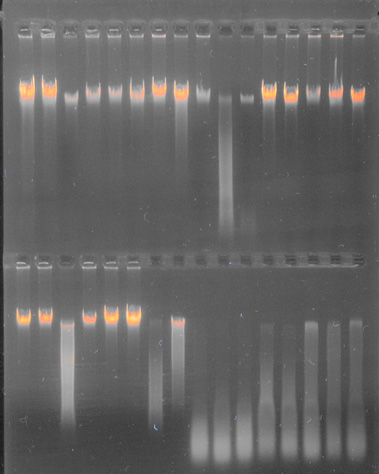
Electroforegrama în gel de agaroză 0,7 %, in 0,5 x TBE, pentru testarea calităţii ADN-ului extras. Probele degradate prezintă dâre alungite datorate fragmentării ADN-ului(foto original R.Suciu)
Analiza ADN-ului prin PCR / secvențiere directă
Reacţia PCR (Polymerase Chain Reaction) reprezintă o tehnică ce permite amplificarea unor fragmente specifice de ADN, aflate în concentraţie foarte mică într-o probă.
Amplificările PCR au fost realizate într-un termociclator GeneAmp PCR System 9700 (AppliedBiosystems).
Pentru amplificarea regiunii D-loop(regiunea de control) din ADNmt (~630 bp) am utilizat perechea de primeri dezvoltată de (Congiu et al. 2011) și aplicată anterior pentru Acipensernaccarii.
Acip PHE 1F, (5′-CACCCTTAACTCCCAAAGC-3′)
Acip PHE 1R (5′- CCCATCTTAACATCTTCAGT-3′)
Probele de ADN extrase de la sturioni, pregătite pentru amplificare PCR în termociclatorul GeneAmp PCR System 9700 (AppliedBiosystems)(foto original R.Suciu)
Amplificările PCR au fost realizate în termociclatorul GeneAmp PCR System 9700 (AppliedBiosystems), în volume de reacţie de 50 μl conținând:
- pentru probele ADN de morun: - 0,4U de GoTaqPolimerază (Promega), 10 μl tampon 5x flexi, 100 μMdNTP, MgCl2 =4ul, 100 μM din fiecare primerşi 200 ng de ADN.
Volumul de reacţie a fost completat cu apă bidistilată sterilă.
Condițiile de reacție pentru morun și nisetru au fost următoarele:
I. etapa inițială - 3 min la 94 ˚C
II. 30 x
30s la 94 ˚C
45s la 53 ˚C
1 min la 72 ˚C
III. elongare finală 5 min la 72 ˚C
Condițiile de reacție pentru PCR la păstrugă au fost următoarele:
I. 3 min la 94 ˚C
II. 30 x
30s la 94 ˚C
45s la 53,5 ˚C
1 min la 72 ˚C
III. 5 min la 72 ˚C (primeri 0,25ul)
După finalizarea reacției PCR produșii de amplificare sunt vizualizați prin electroforeza în gel de agaroză. La finalul migrării, vizualizarea benzilor se realizează cu ajutorul unui transiluminator UV.
Concentrațiile ADN-ului amplificat in reacția PCR au fost citite cu aparatul ScanDrop și apoi au fost pregătite pentru a fi expediate pentru secvențierea la Macrogen Europe / Amsterdam. (www.macrogen.eu )
Analiza datelor
Secvențele de nucleotide au fost aliniate folosind algoritmul Clustal W (BioEdit) și verificate manual. Numărul de haplotipuri și frecvența haplotipurilor a fost calculată utilizând programul Arlequin(Excoffier & Lischer 2010) iar reprezentarea grafică s-a realizat folosind programul Excel / Microsoft Office.
Construcția arborilor de înrudire (filogenetici) prin metoda NeighbourJoining a fost realizată cu programul Mega 6 (Tamura et al. 2013).